[공부 자료 : Must Have 데싸노트의 실전에서 통하는 머신러닝]
# K-최근접 이웃 (KNN, K-Nearest Neighbors)
- K-최근접 이웃
K-최근접 이웃 (KNN, K-Nearest Neighbors)
종속변수가 범주형이며 개수가 3개 이상인 다중 분류(multiclassification)를 다룰 때 사용
데이터 간의 거리를 활용하여 새로운 데이터를 예측하는 모델
KNN의 장점
간단하고 직관적
별도의 가정이 없다 (선형 회귀는 독립변수와 종속변수의 선형 관계를 가정한다)
작은 데이터set에 적합
KNN의 단점
데이터가 커질수록 속도가 느려진다
아웃라이어(평균치에서 크게 벗어나는 데이터)에 취약
- 스케일링
스케일링 : KNN은 거리를 기준(가까운가, 먼가)으로 예측을 하기 때문에 독립변수 사이의 스케일이 중요하다
- 동점 문제
동점 문제 : KNN은 거리를 기준으로 예측을 하기 때문에, 동점이 발생할 경우 판단이 어렵다
n_neighbors : 고려할 이웃의 수를 항상 홀수로 유지한다
weights : 동점일 경우, 거리가 더 가까운 쪽으로 결정한다 (가중치)
- KNN의 특징
KNN 알고리즘의 계산에서, fit( ) 과정에서는 사실 크게 학습하는 것이 없다
선형 회귀, 로지스틱 회귀 : fit( ) 과정에서 수식을 만들어 predict( )에 사용
KNN : fit( )은 단지 스크린샷을 찍는 용도, predict( )에서 실제로 학습 데이터와 시험 데이터 사이의 거리를 계산한다
데이터의 규모가 커지면 predict( )가 오래 걸릴 수 있다
# KNN - 와인 등급 예측하기
- 1. 데이터 불러오기
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns
file_url = 'https://media.githubusercontent.com/media/musthave-ML10/data_source/main/wine.csv'
data = pd.read_csv(file_url)
data.head()- 2. 데이터 분석하기
변수 : 총 13개
독립변수 : 12개
종속변수 : 1개 (cass)
data.columns
# Index(['alcohol', 'malic_acid', 'ash', 'alcalinity_of_ash', 'magnesium',
# 'total_phenols', 'flavanoids', 'nonflavanoid_phenols',
# 'proanthocyanins', 'color_intensity', 'hue',
# 'od280/od315_of_diluted_wines', 'proline', 'class'],
# dtype='object')data.info()
# # Column Non-Null Count Dtype
# --- ------ -------------- -----
# 0 alcohol 176 non-null float64 - 결측치 2개
# 1 malic_acid 178 non-null float64
# 2 ash 178 non-null float64
# 3 alcalinity_of_ash 178 non-null float64
# 4 magnesium 178 non-null int64
# 5 total_phenols 178 non-null float64
# 6 flavanoids 178 non-null float64
# 7 nonflavanoid_phenols 173 non-null float64 - 결측치 5개
# 8 proanthocyanins 178 non-null float64
# 9 color_intensity 178 non-null float64
# 10 hue 178 non-null float64
# 11 od280/od315_of_diluted_wines 178 non-null float64
# 12 proline 178 non-null int64
# 13 class 178 non-null int64data.describe()
(1) min-max 비교 : 변수마다 값의 범위가 상당히 다르다
변수의 스케일이 다르다 → 스케일링(scaling, 독립변수의 범위를 동일한 수준으로 맞추는 작업) 필요
(거리 기반 알고리즘이 아니라면 굳이 안해도 된다!)
(2) min-25%-50%-75%-max 변화에서 값이 갑자기 급증한다 (color_intensity) : 아웃라이어 존재
- 3. 종속변수 분석하기
sns.barpot(x, y) : 막대 그래프
sns.countplot(data) : 데이터에 대한 막대 그래프
data['class'].unique() # 고유값
# array([0, 1, 2]) - 종속변수가 3개의 클래스를 갖는 다중 분류
data['class'].nunique() # 고유값의 개수
# 3
data['class'].value_counts() # 고유값이 각각 몇 개씩
# 1 71
# 0 59
# 2 48
# Name: class, dtype: int64
sns.barplot(x = data['class'].value_counts().index, y = data['class'].value_counts())
# .index : data['class'].value_counts에서 index만 뽑아내기
# Seaborn에서는 동일 기능을 함수로 제공
sns.countplot(data['class'])
- 4-1. 데이터 전처리 - 결측치 처리하기
isna( ) : 결측치 확인하기
data.isna() # null에 해당하는 요소를 True로 반환 (비효율)
data.isna().sum()
# alcohol 2 : 결측치 2개
# malic_acid 0
# ash 0
# alcalinity_of_ash 0
# magnesium 0
# total_phenols 0
# flavanoids 0
# nonflavanoid_phenols 5 : 결측치 5개
# proanthocyanins 0
# color_intensity 0
# hue 0
# od280/od315_of_diluted_wines 0
# proline 0
# class 0
# dtype: int64
data.isna().mean()
# alcohol 0.011236
# malic_acid 0.000000
# ash 0.000000
# alcalinity_of_ash 0.000000
# magnesium 0.000000
# total_phenols 0.000000
# flavanoids 0.000000
# nonflavanoid_phenols 0.028090
# proanthocyanins 0.000000
# color_intensity 0.000000
# hue 0.000000
# od280/od315_of_diluted_wines 0.000000
# proline 0.000000
# class 0.000000
# dtype: float64dropna( ) : 결측치가 있는 행 전체를 제거하기 (원본 data는 변하지 않는다)
data.dropna()
# row : 178 -> 171
data.dropna().isna().sum()
# alcohol 0
# malic_acid 0
# ash 0
# alcalinity_of_ash 0
# magnesium 0
# total_phenols 0
# flavanoids 0
# nonflavanoid_phenols 0
# proanthocyanins 0
# color_intensity 0
# hue 0
# od280/od315_of_diluted_wines 0
# proline 0
# class 0
# dtype: int64
# 아직 데이터 원본의 결측치는 처리되지 않았다
# 데이터 원본의 결측치가 들어있는 행을 삭제하는 방법
# 1. data = data.dropna()
# 2. data.dropna(inplace = True)
# 원하는 특정 변수의 결측치만 처리하기
data.dropna(subset = ['alcohol'])
# rows : 178 -> 176drop(col명, axis = 1) : 결측치를 갖고 있는 변수(col) 자체를 제거하기 (원본 data는 변하지 않는다)
data.drop(['alcohol', 'nonflavanoid_phenols'], axis = 1)
# 원본 데이터에는 영향을 주지 않는다fillna( ) : 결측치 채우기 (원본 data는 변하지 않는다)
data.fillna(100)
# 결측치를 99로 채우기
data.fillna(data.mean())
# 각 col의 평균으로 결측치를 채우기
data.fillna(data.median())
# 각 col의 중위값으로 결측치를 채우기
data.fillna(method = 'bfill')
# 결측치를 바로 아랫값과 동일하게 채우기
data.fillna(method = 'ffill')
# 결측치를 바로 윗값과 동일하게 채우기1) 일반적으로 결측치는 dropna( )를 사용하여 row 자체를 지운다
어떠한 방식으로 결측치를 채우든, 실제값과 일치할 가능성이 낮기 때문에 노이즈로 작용할 가능성이 높기 때문
dropna( )의 문제점 : 결측치가 많을 경우 너무 많은 데이터(row)가 사라질 수 있다
2) 특정 변수에 결측치가 너무 많을 경우, 차라리 drop( )을 통해 변수 자체를 지워버리는 것이 낫다
특정 변수의 결측치가 50% 이상일 경우 drop( )을 고려
특정 변수의 결측치가 70% 이상일 경우 가급적 drop( )을 사용하는 것이 낫다
3) fillna( )를 사용할 경우, 평균 또는 중위값이 권장된다
inplace = True : 원본 데이터에 덮어쓰기
data.fillna(data.median(), inplace = True)
# 원본 데이터 덮어쓰기- 4-2. 데이터 전처리 - 스케일링
스케일링 : 데이터의 스케일을 맞추는 작업
KNN은 거리 기반 알고리즘
데이터의 각 col마다 값의 범위가 다를 경우, 같은 거리 1이 col마다 차지하는 의미가 달라진다
스케일링 종류
- 표준화 스케일링 : 평균이 0, 표준편차가 1이 되도록 데이터를 고르게 분포
- 로버스트 스케일링 : 데이터에 아웃라이어가 존재하고, 아웃라이어의 영향력을 줄이고 싶을 때
- 최소-최대 스케일링 : 데이터 분포의 특성을 최대한 그래도 유지하고 싶을 때 사용
- 정규화 스케일링 : 행 기준의 스케일링이 필요할 때 사용 (거의 사용하지 X)
from sklearn.preprocessing import StandardScaler, RobustScaler, MinMaxScaler(1) 표준화 스케일링 방법 : 각 데이터 (xi) 에 해당 데이터가 포함된 변수 (x) 의 평균을 빼고, 표준편차로 나눈다
모든 col이 표준정규분포의 형태를 따른다
fit( ) : 각 col의 평균과 표준편차를 구한다
transform( ) : 공식을 적용하여 표준정규분포 형태 (평균 = 0, 표준편차 = 1) 를 만든다

스케일링 적용 순서
스케일러 선정 → scaler.fit(data) → scaled data = scaler.transform(data) → pd.DataFrame(scaled data)
스케일러 선정 → scaled data = scaler.fit_tranform(data) → pd.DataFrame(scaled data)
# 표준화 스케일링
st_scaler = StandardScaler() # 스케일러 지정
st_scaler.fit(data) # 학습
st_scaled = st_scaler.transform(data) # 스케일링 적용
st_scaled # Numpy Array (컴퓨터가 보기 좋다)
pd.DataFrame(st_scaled) # 변형된 데이터에는 col명이 빠져있다
st_scaled = pd.DataFrame(st_scaled, columns = data.columns) # col명 추가
st_scaled
round(st_scaled.describe(), 2) # 모든 변수의 평균이 0, 표준편차가 1(2) 로버스트 스케일링 : 각 변수의 평균과 표준편차 대신 사분위값(Q1 - 25%, Q2 - 50%, Q3 - 75%)을 이용
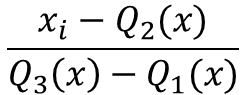
# 로버스트 스케일링
rb_scaler = RobustScaler() # 로버스트 스케일링에 사용될 객체 생성
rb_scaled = rb_scaler.fit_transform(data) # fit_transform = fit + transform
rb_scaled = pd.DataFrame(rb_scaled, columns = data.columns)
round(rb_scaled.describe(), 2)(3) 최소-최대 스케일링 : 각 변수의 최대값이 1, 최소값이 0이 되도록 변형

mm_scaler = MinMaxScaler() # 최소-최대 스케일링 객체 생성
mm_scaled = mm_scaler.fit_transform(data)
mm_scaled = pd.DataFrame(mm_scaled, columns = data.columns)
round(mm_scaled.describe(), 2) # 모든 변수의 최대값이 1, 최소값이 0어떤 스케일링 방식을 선택해야 하는가?
1) 표준화 스케일링
아웃라이어가 존재할 때, 아웃라이어의 영향을 받는다 (아웃라이어에 취약)
데이터의 기존 분포 형태가 사라지고 정규분포를 따르는 결과물을 얻는다
가장 무난
→ 데이터의 분포가 정규분포와 상당히 거리가 있으면 기존 데이터 특성을 잃을 수 있다
→ 아웃라이어가 상당수 있을 때는 기존 데이터 특성을 잃을 수 있다
2) 로버스트 스케일링
아웃라이어가 존재할 때, 아웃라이어의 영향을 받지 않는다
→ 데이터가 아웃라이어의 영향을 크게 받을 때, 이를 피하고 싶다면 로버스트 스케일링을 사용
변형된 데이터의 범위가 표준화/최소-최대 스케일링보다 넓게 나타난다
3) 최소-최대 스케일링
아웃라이어가 존재할 때, 아웃라이어의 영향을 받는다 (아웃라이어에 취약)
표준화/로버스트 스케일링보다 기존 데이터의 분포를 가장 있는 그대로 담아낸다
→ 기존 데이터의 분포를 최대한 유지하고 싶을 때 최소-최대 스케일링을 사용
스케일링 주의사항
스케일링 학습(fit( ))에는 X_train만 사용해야 한다
스케일링 변환(transform( ))에는 X_train과 X_test를 해줘야 한다
from sklearn.model_selection import train_test_split
# 독립변수와 종속변수
X = data.drop('class', axis = 1)
y = data['class']
# 훈련셋과 시험셋
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size = 0.2, random_state = 100)
# 최소-최대 스케일링 사용
from sklearn.preprocessing import MinMaxScaler
mm_scaler = MinMaxScaler()
# 스케일링 학습
mm_scaler.fit(X_train)
# 스케일링 변환
X_train_scaled = mm_scaler.transform(X_train)
X_test_scaled = mm_scaler.transform(X_test)
# X_train_scaeld = mm_scaler.fit_tranform(X_train)
# X_test_scaeld = mm_scaler.fit_tranform(X_test)- 5. 모델링 - KNN classifer
from sklearn.neighbors import KNeighborsClassifier
knn = KNeighborsClassifier() # KNN 분류 모델 생성
knn.fit(X_train_scaled, y_train) # 학습 (스케일링 된 데이터를 이용)- 6. 예측
pred = knn.predict(X_test_scaled) # 예측 (스케일링 된 데이터를 이용)
pred
# array([1, 2, 0, 1, 2, 2, 1, 2, 1, 0, 2, 0, 2, 2, 2, 0, 2, 0, 1, 0, 2, 0,
# 2, 1, 0, 0, 1, 1, 1, 2, 2, 1, 0, 1, 2, 2])- 7. 평가
from sklearn.metrics import accuracy_score
accuracy_score(y_test, pred)
# 0.8888888888888888- 8. KNN - 하이퍼 파라미터 튜닝
KNeighborsClassifier, KNeighborsRegression 주요 파라미터
n_neighbors : 예측에 참고할 이웃 수
default = 5
weights : 예측에 사용되는 가중치
default = 'uniform' : 모든 포인트에 동일한 가중치
'distance' : 사용자 정의 함수
metric : 거리 측정 기준
default = 'mnkowski'
n_jobs : 실행할 병렬 작업 수
default = None
# n_neighbors를 1부터 20까지 넣어서 결과 비교하기
for i in range(1, 21):
knn = KNeighborsClassifier(n_neighbors = i)
knn.fit(X_train_scaled, y_train)
pred = knn.predict(X_test_scaled)
print('n_neighbors = {}일 때 accuracy_score : {}'.format(i, round(accuracy_score(y_test, pred), 4)))
# n_neighbors = 1일 때 accuracy_score : 0.9167
# n_neighbors = 2일 때 accuracy_score : 0.8889
# n_neighbors = 3일 때 accuracy_score : 0.8889
# n_neighbors = 4일 때 accuracy_score : 0.9167
# n_neighbors = 5일 때 accuracy_score : 0.8889
# n_neighbors = 6일 때 accuracy_score : 0.9167
# n_neighbors = 7일 때 accuracy_score : 0.9167
# n_neighbors = 8일 때 accuracy_score : 0.8889
# n_neighbors = 9일 때 accuracy_score : 0.8889
# n_neighbors = 10일 때 accuracy_score : 0.8889
# n_neighbors = 11일 때 accuracy_score : 0.9167
# n_neighbors = 12일 때 accuracy_score : 0.9167
# n_neighbors = 13일 때 accuracy_score : 0.9722
# n_neighbors = 14일 때 accuracy_score : 0.9444
# n_neighbors = 15일 때 accuracy_score : 0.9444
# n_neighbors = 16일 때 accuracy_score : 0.9444
# n_neighbors = 17일 때 accuracy_score : 0.9167
# n_neighbors = 18일 때 accuracy_score : 0.9722
# n_neighbors = 19일 때 accuracy_score : 0.9444
# n_neighbors = 20일 때 accuracy_score : 0.9722선형 그래프로 그리기
scores = []
for i in range(1, 21):
knn = KNeighborsClassifier(n_neighbors = i)
knn.fit(X_train_scaled, y_train)
pred = knn.predict(X_test_scaled)
acc = accuracy_score(y_test, pred)
scores.append(acc)
import matplotlib.pyplot as plt
import seaborn as sns
# 선형 그래프 그리기
sns.lineplot(x = range(1, 21), y = scores)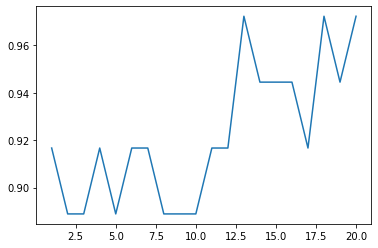
좋은 매개변수 선정 : 정확도는 높으면서도 연산 횟수는 적은 값 =13
'머신러닝' 카테고리의 다른 글
| 08. 지도학습 (분류, 회귀) - 결정 트리 : 연봉 수준 예측하기 (0) | 2023.01.25 |
|---|---|
| 07. 지도 학습 (분류) - 나이브 베이즈 : 스팸 메일 분류하기 (0) | 2023.01.25 |
| 05. 지도 학습 (분류) - 로지스틱 회귀 : 타이타닉 생존자 예측하기 (0) | 2023.01.21 |
| 04. 지도 학습 (회귀) - 선형회귀 : 보험료 예측하기 (0) | 2023.01.21 |
| 03. Numpy (0) | 2023.01.14 |



